Des scientifiques du Wyss Institute for Biologically Inspired Engineering d’Harvard ont créé un outil d’édition génomique, soit une technique de manipulation du génome [ensemble du matériel génétique d’une espèce codé dans son ADN] visant à la modification du matériel génétique. L’avancée baptisée Retron Library Recombineering (RLR) vient répondre à certaines problématiques de CRISPR, la technique d’édition génomique actuellement la plus reconnue et qui a fait plusieurs fois la une, notamment à la suite de son utilisation pour modifier l’ADN d’embryons humains.
La RLR s’appuie sur les retrons, soit des séquences génétiques présentes dans certaines bactéries qui produisent un brin d’ADN unique. Vulgairement, la RLR consiste à introduire dans une cellule une mutation sous la forme de brin d’ADN pendant qu’elle est dans la phase de réplique de son génome. Par la suite, les cellules sœurs créées répliquent naturellement la mutation. « La RLR est un outil d’édition génétique plus simple et plus flexible qui peut être utilisé pour des expériences hautement multiplexées », soutient Max Schubert, l’un des coauteurs de l’étude.
Inscrivez-vous à la newsletter
En vous inscrivant vous acceptez notre politique de protection des données personnelles.
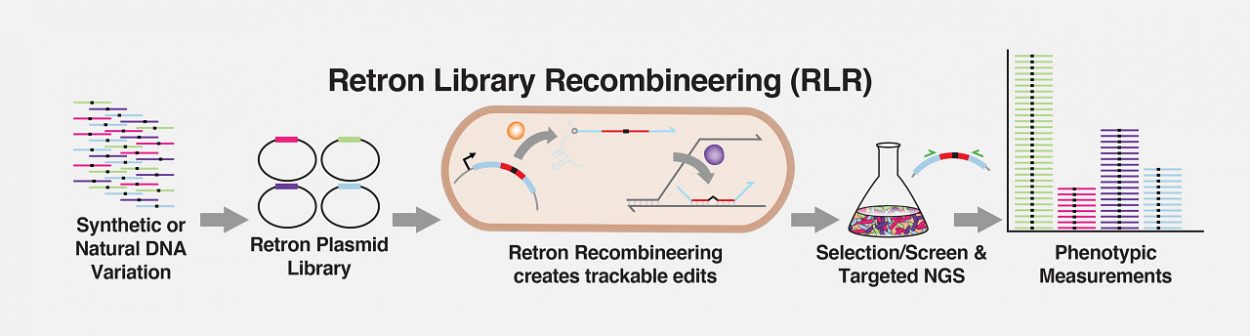
Les retrons permettent la production et la sélection rapides de millions de variations d’ADN traçables et de leurs conséquences sur les bactéries en simultané. Crédit : Max Schubert / Institut Wyss de l’université de Harvard
La technique actuelle, CRISPR, permet de modifier les séquences d’ADN grâce à des « ciseaux génétiques » : l’enzyme Cas9. Simplement, CRISPR permet d’ajouter, supprimer ou modifier le patrimoine génétique de n’importe quelle cellule. La technique s’est montrée prometteuse, notamment pour la recherche contre le cancer. Néanmoins, le matériel requis pour CRISPR est difficile à installer en grande quantité, ce qui complexifie la recherche. Par ailleurs, l’enzyme Cas9 sur lequel CRISPR s’appuie pour couper les brins d’ADN n’est pas toujours précis et peut couper des éléments non-ciblés.
Contrairement à CRISPR, avec la RLR, l’ADN n’est pas cassé, ce qui supprime « la toxicité souvent observée avec CRISPR ». Aussi, les retrons font office de « code-barre » et permettent une identification. Ainsi, les chercheurs peuvent faire de l’édition génomique sur plusieurs cibles dans lesquelles ils veulent insérer des mutations. La RLR pourra être utilisée conjointement avec CRISPR ou servir de remplacement quand le découpage est trop délicat.
La RLR peut « conduire à des innovations nouvelles, passionnantes et inattendues »
« Nous avons pensé que les retrons devraient nous donner la capacité de produire de l’ADN simple brin à l’intérieur des cellules que nous voulions modifier, plutôt que d’essayer de les forcer à entrer dans la cellule de l’extérieur, et sans endommager l’ADN natif, deux qualités très convaincantes », explique Daniel Goodman, ancien doctorant de l’Institut Wyss.
Le groupe de recherche d’Harvard a testé la RLR sur des bactéries E. coli et ont observé que 90% de la population incorporait la séquence après quelques modifications. Pendant leurs expériences, les scientifiques ont pu trouver des mutations de résistance aux antibiotiques au sein de la bactérie E. coli en séquençant le « code-barre » des retrons – au lieu de séquencer les mutants – rendant ainsi la procédure nettement plus rapide.
« La RLR nous a permis de faire quelque chose d’impossible avec CRISPR : nous avons découpé aléatoirement un génome bactérien, nous avons transformé ces fragments génétiques en ADN simple brin in situ, et nous les avons utilisés pour cribler des millions de séquences simultanément », détaille Max Schubert.
Malgré ses avancées encourageantes, il reste du chemin à parcourir avant que la RLR soit utilisée à grande échelle, notamment sur l’amélioration et la standardisation de son taux d’édition. Néanmoins, l’équipe du Wyss Institute estime qu’elle peut « conduire à des innovations nouvelles, passionnantes et inattendues ».
